研究内容
当研究室では、生物進化の歴史とメカニズムの解明に、分子的アプローチで取り組んでいます。これまで主に爬虫類や淡水魚類などの脊椎動物を研究材料にしてきましたが、必ずしもそれに限定する訳ではありません。当研究室の研究プロジェクトについて以下に簡単に説明します。
(1)分子系統学による生物進化史の解明
生物進化の歴史は、現生生物のゲノムDNAの中に乱雑に重ね書きされて記録されています。進化的に有用な分子データを実験やデータマイニングによって取得し、コンピュータを用いて解析することによって、種や遺伝子の系統関係(枝分かれ進化のパターンや分岐した年代)を解明します。また、それを他の情報(形態進化、化石情報、大陸移動、古気候など)と対比させ、生物進化を引き起こした環境要因を探ります。 分子データとしては、ミトコンドリアゲノム全塩基配列や核ゲノムコードの遺伝子群のほか、最近では次世代シーケンサー(図1)を用いて取得したRNA-Seqデータなども用いています。
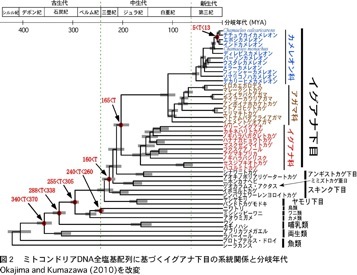
我々は大陸移動に伴って分断的に系統分岐した淡水魚や爬虫類のグループをいくつか同定しました。例えばイグアナ下目の系統関係(図2)に基づき、ゴンドワナ超大陸起源のアガマ科が大陸移動に伴ってユーラシア大陸に渡来したとの仮説を提示しました。他の研究例に興味がある方は次のリンク先をクリックして下さい。
●日本語解説文「トカゲ類の高次系統関係」「トゲオアガマ類の進化」「タイワンドジョウ類の生物地理」
(2)ゲノムや遺伝子の分子進化の解明
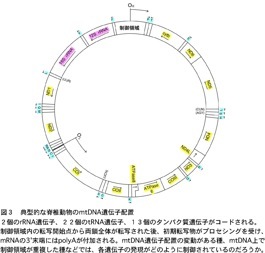
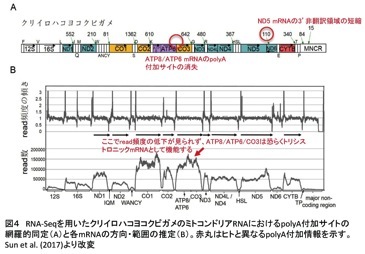
当研究室では、ミトコンドリアゲノムや核ゲノムにコードされる特定の遺伝子に注目し、それらの発現様式及び分子進化様式を解明する研究を行っています。脊椎動物のミトコンドリアゲノムには37個の遺伝子がコードされています(図3)。RNA-Seq解析によってそれらの転写物を網羅的に調べることにより、ミトコンドリアRNAのポリアデニル化サイトのゲノムワイドな同定や、ミトコンドリアmRNAの方向・範囲などの推定ができることを示しました。この方法を用いてミトコンドリアmRNAの多様性と進化に関する研究を行っています(図4)。他に嗅覚受容体遺伝子の分子進化の研究なども行っています。
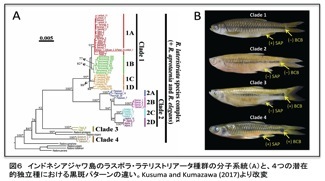
この他にも、保全対策を要する種や集団(インドネシアの淡水魚、名古屋市内のサンショウウオなど)について、その遺伝的多様性を解析する研究も行っています(図6)。共同研究者は、国内のみならずタイ、ベトナム、エジプトなどにも広がっており、国際的な研究展開を目指しています。
研究室の詳細は、以下のURLを御覧下さい。http://www.nsc.nagoya-cu.ac.jp/〜kuma/index.html
各図をクリックすると大きな画面が開きます
(3)東海地方の動物の種多様性と遺伝的多様性に関する研究
この研究は、研究科附属の生物多様性研究センターと連携して行っています。名古屋市を中心とした東海地方に生息する動物(主に貝類や昆虫類)のサンプルを収集し、ミトコンドリアDNAの特定領域の塩基配列を決定して分析するDNAバーコーディングの研究を行っています。DNA塩基配列は種内の個体間ではほぼ同じですが、種間では多くの場合2%以上異なります。DNAバーコードデータベースは、形態情報とは別に種同定を行うための有力な情報源となります。分類学などの学術分野にとどまらず、環境保全、疫学、食品管理など様々な目的で利益をもたらすことが期待されています。
昆虫類についてDNAバーコーディング研究を進めてみると、意外なことも分かりました。愛知県に生息するニセチビヒョウタンゾウムシは形態的に似通っているため単一種と考えられてきましたが、DNAバーコーディングの結果からは、山一つ隔てると別種に分かれている可能性が示されました(図5)(新聞記事)。