関連研究
ペプチド鎖伸長因子EF-1αでみた真核生物の進化と分類(1996)
はじめに
分子系統樹は、特定遺伝子の塩基配列や特定タンパク質のアミノ酸配列にみられる生物間の置換率をもとに描かれる生物の系統樹で、分子サイズが小さく解析の容易な5SrRNAやシトクロームcを用いて数多くの分子系統樹が描かれてきた。そして、それらは古細菌の分類や位置づけ、各種生物の進化的な位置関係等、化石資料や形態学的な分類では推測できない多くの事実を明らかにし、分子系統樹の地位を築き上げてきた。しかし、5SrRNAやシトクロームcは、分子サイズが小さいが故に生じる誤差が大きく、信頼性に欠けるという指摘もあり、現在では、より分子サイズの大きなRNAやタンパク質に研究の対象が移っている。EF-1αも優れた分子時計であることから、統計数理研究所の長谷川氏らを中心に解析が行われ、これまでに古細菌の進化的な位置づけや真核生物の初期進化に関する知見が報告(1)されている。しかし、報告された分子系統樹は、比較されている生物の種類が少なく、真核生物の詳細な進化過程を読みとることはできない。また、分子系統樹は、解析方法により異なる結果を示すこともある。よって、本研究では、筆者らが明らかにしたイネ(Oryza sativa)の EF-1αも含め、54種類の生物のデータを活用し、ギャップ領域を考慮に入れたEF-1αの分子系統樹を作成した。
解析方法
EF-1αのアミノ酸配列は、SWISS-PROT(アミノ酸配列のデータバンク)で検索し、筆者が明らかにしたイネ(Oryza sativa)を含む54種類の生物の配列データを得た。系統樹の作成には国立遺伝研究所の分子進化学的解析システム(ODEN)を使用した。まず、各アミノ酸配列のマルチプルアライメントを malignで作成し、すべての生物で比較することのできる(相同性が確認できる)424 座位(イネのアミノ酸配列で 5 - 429 番目のアミノ酸)を選択した。つぎに、選択領域のアミノ酸の置換数を dista で推定し、dmata で距離行列を作成した。系統樹の作成には treeupg (UPGM法)を使用した。次に、その結果をグラフィックスとして表現するためソフトウェア Tree Draw Deck ( Felsenstein 氏により作成された分子系統樹を描くためのソフトウェアを Gilbert 氏が改良したもの)に入力し、古細菌をアウトグループとした真核生物の有根分子系統樹を作成した。
結果および考察
【真核生物の初期進化】
最初の真核生物は、単細胞性の原生生物であったことが様々な証拠から明らかにされているが、どの様なタイプの原生生物であったかに関しては明確にされておらず、大きな問題として残っている。分子系統樹は、このような問題を解きあかす上で重要な糸口を与えてくれる。たとえば、図1の分子系統樹をみると、真核生物の根(共通の祖先)より最初に分岐しているのはマラリア病原虫(Entamoeba histolytica)や赤痢アメーバー(Plasmodium falciparum)の様な寄生性の原生生物である。よって、最初の真核生物は寄生性の原生生物に近いものであったことが推察できる。この点については、すでに他の分子系統樹(3,4) でも示唆されいる。
真核生物の初期進化に関する大変興味深い問題に、ミトコンドリアが共生した時期がある。長谷川氏らの分子系統樹ではミトコンドリアを持たない赤痢アメーバーがミトコンドリアを持つマラリア病原虫より先に分岐し、その分岐点を境にミトコンドリアを持つ生物と持たない生物にきれいに分けることができる。よって、ミトコンドリアの共生は、赤痢アメーバーが分岐した後で起こった可能性のあることを示唆している。しかし、図1の分子系統樹では、ミトコンドリアを持つマラリア病原虫がミトコンドリアを持たない赤痢アメーバーより早い時期に分岐しており、ミトコンドリアが共生したのはこのような寄生性の原生生物ではなく、もっと古い時期に存在した原生生物ということになる。長谷川氏との結果に違いが生じたのは、比較したEF-1αのアミノ配列領域(長谷川氏は380座位)および解析方法(長谷川氏はPROTML法)の違いによるものと考えられるが、本研究の結果が正しいとすれば、赤痢アメーバーは、もともとミトコンドリアを持っていたのに、共生という特殊な生活をおくっている間にミトコンドリアを必要としなくなり、進化の過程で失ったことになる。ミトコンドリアを持っている寄生性の原生生物の中には、持ってはいるが機能の退化がみられるものもあり、ミトコンドリアの共生時期に関しては、さらに多くの原生生物を取り込んだ分子系統樹を作成し、解析する必要があるものと思われる。
【菌類、動物、植物の分岐】
真核生物は、形態分類学的に原生生物、菌類、動物、植物に分けられている。では、それらが共通の真核生物の祖先からどの様に分岐し、進化したかという点が問題となる。化石等の資料が少なく、形態学的な判断も困難であることから、長い間、原生生物から菌類、動物、植物がそれぞれ独立に進化してきたと考えられてきた。しかし、分子進化学的な解析が進められた結果、ある種の原生生物から植物が最初に分岐し、その後、菌類と動物に分かれて進化したと推定される結果が蓄積しつつある。図1の分子系統樹からも同様の結果が読みとれる。
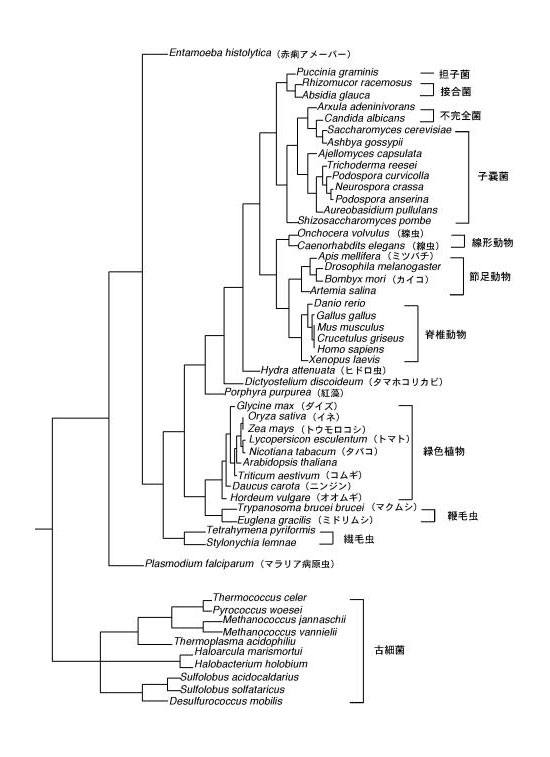
緑色植物や一部の植物性原生生物に存在する葉緑体は、藍藻のような細菌が共生したものであることは明らかにされているが、その時期に関しては不明な点が多い。しかし、図1の分子系統樹は、繊毛虫が分岐した後の原生生物に葉緑体が共生し、一部は単細胞のままミドリムシ(Euglena gracilis )のような葉緑体を持つ植物性鞭毛虫に進化し、一部は多細胞化して緑色植物に進化したことを示している。緑色植物とミドリムシが一つの生物から分岐している点も興味深い。高等植物の分類が形態学的な分類と大きく異なっているが、これは、緑色植物間の相同性が非常に高く、わずかな差が大きく反映されてしまった結果であると考えられる。また、動物性鞭毛虫のマクムシ(Trypanosoma brucei brucei)が植物と近い位置に分類されているが、この生物は、進化の過程で葉緑体を欠失したミドリムシの仲間であることが示唆されているので矛盾した結果ではない。
動物と菌類に至る進化に関しては、長谷川氏らの分子系統樹とは若干異なる結果を示していた。まず、細胞性粘菌であるタマホコリカビ(Dictyostelium discoideum)の分岐が、植物が分岐した後の動物と菌類に共通な祖先生物から起きていた。この原生生物は、環境が整うとアメーバーのような形でバクテリアを捕獲し消化することで生きているが、環境が悪化すると凝集してナメクジの様になり場所を移動する。さらには、子実体に変態して胞子を形成し、休眠にはいることもある。そんな動物と菌類の両方の特徴を備えている生物が、植物が分岐した後の動物と菌類に共通な祖先生物から分岐しており、動物と菌類の祖先がこのような生物であった可能性を示唆している。さらに、図1の分子系統樹からは、菌類に至った原生生物が担子菌や接合菌のグループと不完全菌や子嚢菌のグループに独立に進化したことや、動物に至った原生生物からは線虫のような線形動物が最初に分岐し、その後節足動物と脊椎動物が分岐したこと等の進化の過程を読みとることができる。また、単細胞生物から多細胞生物への進化が数カ所(緑色植物の祖先が鞭毛虫と分岐した後、動物の祖先がタマホコリカビと分岐した後、等)で起こったことを示している点も興味深い。
【ギャップ領域でみた生物の分類】
以上のように、EF-1αは分子系統樹を作成する優れた分子時計であることが確認された。しかし、分子系統樹のコンピューター解析プログラムの作成には多くの仮定が含まれ、解析方法によっては違った分類に属することもある。そこで本研究では、植物、菌類、および動物の進化的位置づけに関するより直接的な情報を得るため、系統樹の作成時に malign で作成したマルチプルアライメントを活用し、その中に存在する特徴的なギャップ領域に着目して生物の進化および分類に関する検討を行なった。図2は、ギャップ領域のアミノ酸配列を示している。興味深いことに、真核生物はギャップを持つ生物(原生生物と植物)と持たない生物(菌類と動物)に分類することができる。菌類と動物のみにアミノ酸配列の挿入がみられ、その相同性が非常に高いことから、アミノ酸配列に対応するDNAの挿入が起きたのは、植物が分岐した後の菌類と動物に共通な祖先ということになる。図1の分子系統樹と照らし合わせてみるとタマホコリカビのような生物からヒドロ虫のような生物に至る過程で長いDNAの挿入がEF-1α遺伝子上に起きたことになる。現在、EF-1αのアミノ酸配列にギャップを持つ原生生物は確認されていないが、そのような原生生物が見つかれば、菌類と動物に共通な祖先の形が見えてくるものと思われる。

最後に
本研究で作成したEF-1αの分子系統樹は、緑色植物と鞭毛虫が共通の祖先から分岐していることや菌類と動物の祖先が細胞性粘菌に類似した生物であった可能性のあること等、大変興味深い結果を示していた。しかし、イネをはじめ、アラビドプシス(3) やアフリカツメガエル(4) で、EF-1αの遺伝子が重複していることを考えると、分子系統樹の作成および解釈を慎重に行わなければならない。重複した遺伝子がある場合には、EF-1αとしての機能的制約が軽減し、もとのEF-1αと同じ進化速度を持たなくなることが考えられので、系統樹の作成に用いているEF-1αの遺伝子が、重複したEF-1α遺伝子であった場合、系統樹上での位置がずれてしまう可能性があるからである。実際に、アフリカツメガエルには3種類の重複したEF-1αの遺伝子があるが、それぞれのアミノ酸配列が異なり、用いる分子種によっては脊椎動物以外に分類されてしまう。分子系統樹を描く場合には、この様な可能性があることに注意しなければならない。
参考文献
1. Nakamura, Y et al. : Phylogenetic position of kinetoplastid protozoa inferred from protein phylogenies of elongation factor EF-1a. J.Biochem 119 : 70-79 (1996).
2. Hori, H et al. : Evolutionary change in 5S rRNA secondary structure and a phylogenic tree of 352 5S rRNA species. Biosystems 19(3), 163-172 (1986).
3. Axelos,M et al. : The gene family encoding the Arabidopsis thaliana translation elongation factor EF-1a : Molecular cloning, characterization and expression. Mol Gen Genet 219 : 106-112 (1989).
4. Die,MK et al. : Three genes under different developmental control encode elongation factor EF-1a. Nucl Acids Res 18 : 3489-3493 (1990).
 HOME
HOME 研究室
研究室 遺伝子解析ツール
遺伝子解析ツール 活動報告
活動報告