高等植物の無細胞遺伝子解析系
高等植物の in vitro 転写系
植物培養細胞(用語解説参照)を使って、植物細胞の核で進行する転写(RNA合成)活性を無細胞環境 (in vitro) で解析しています。この方法は、分子のレベルで詳細に遺伝子発現機構を調べる有効な手段です。しかし、これまで植物の分野では技術的に困難であったため広く用いられていません。本研究室では独自の技術で困難を克服し、解析を進めています。
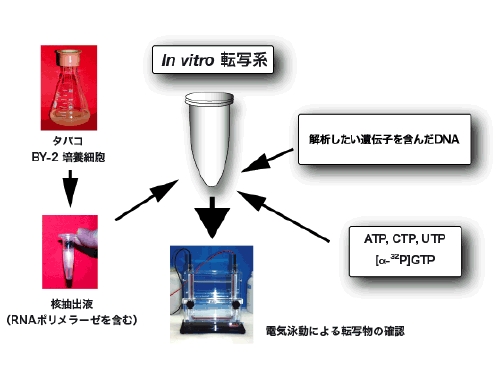
in vitro転写系の解析原理
植物細胞の核には、現在5つの RNA ポリメラーゼの存在が確認され、RNAが合成(転写)されています。RNA ポリメラーゼ I (Pol I) はリボソーム RNA (rRNA) 、RNA ポリメラーゼ II (Pol II) はメッセンジャー RNA (mRNA)と低分子 RNAを転写します。そして RNA ポリメラーゼ III (Pol III)は tRNAや 5S rRNAといった遺伝子発現に関わる重要な安定低分子 RNA を大量に合成します。RNA ポリメラーゼ IV (Pol IV) とRNA ポリメラーゼ V (Pol V) は短いRNAを合成して、遺伝子の抑制機構(サイレンシング)に働くと考えられています。
|
ポリメラーゼ
|
転写する遺伝子、役割など
|
|---|---|
|
Pol I
|
リボソームRNA(rRNA)遺伝子、リボソームの構成要素合成
|
|
Pol II
|
mRNA、低分子RNA
|
|
Pol III
|
tRNA、5S rRNA、核内低分子RNA(snRNA)遺伝子など
|
|
Pol IV
|
siRNAを合成、RNA依存的DNAメチル化に関与、植物特有
|
|
Pol V
|
RNA依存的DNAメチル化反応の足場となるRNA合成に関与、植物特有
|
これらのうち、タバコ培養細胞由来の in vitro 転写系は Pol I ~ III の転写活性を持っています。当研究室ではその中で最も高い転写活性を示す Pol III に特に着目して、研究を進めています。
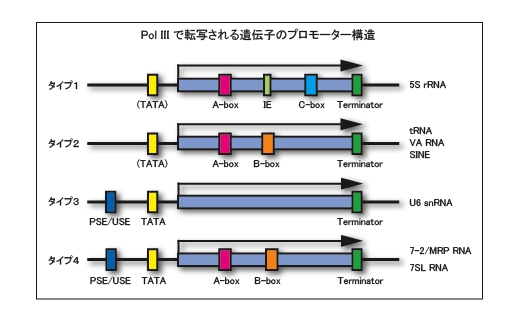
RNAポリメラーゼIIIによって転写される遺伝子の構造
植物の遺伝子発現制御機構は、動物や菌類とは異なった特徴があると考えられています。したがって、植物特有の転写の仕組みを理解することで初めて、真核細胞の転写機構を完全に解明することができます。現在開発を進めているシロイヌナズナ MM2d 細胞由来の in vitro 系は Pol II の活性が強くmRNA 転写の研究に適しています。
tRNAの細胞内での発現量を蛍光タンパク質で検出する方法の論文が、2019年のGene誌
に掲載されました。
植物遺伝子の光依存転写制御に関する論文が、2016年のPlant Physiology
に掲載されました
tRNA 遺伝子の転写制御に関する論文が、2013年のGene誌
に掲載されました
tRNA 遺伝子の転写制御に関する論文が、2011年のGenomics誌
に掲載されました
in vitro 翻訳系
試験管内(in vitro)で合成したタンパク質を電気泳動で確認します。 例えば、葉緑体は植物の細胞の中にある細胞小器官であり光合成を行う場所です。光合成とは、太陽の光エネルギーを利用して、空気中の二酸化炭素から有機物を合成する葉緑体の働きです。植物の葉緑体は光合成を進めるために様々なタンパク質を作り続ける必要があります。その一つであるルビスコ(リブロース1,5ビスリン酸カルボキシラーゼ/オキシダーゼ)は葉緑体の重量のおよそ半分を占め、地球上で最も存在量の多いタンパク質の一つとして知られています。つまり、植物は優れたタンパク質合成の潜在力が備わっていることになります。
現在は基礎研究として、培養細胞の細胞質由来 in vitro(無細胞)翻訳系を使って解析を進めています。開発した in vitro 翻訳系は放射性同位元素を用いる代わりに、緑色蛍光タンパク質(GFP)を使用することで高速・高感度で翻訳活性を解析できます。
葉緑体 psbD-psbC mRNA の翻訳の研究が、2012年のNucleic Acids Research誌
に掲載されました
葉緑体 atpB-atpE mRNA の翻訳の研究が、2011年のNucleic Acids Research誌
に掲載されました





