 DNAバーコード計画
DNAバーコード計画
DNAバーコード計画とは
計画概要
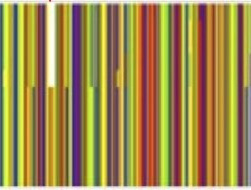 DNA塩基配列を生物固有のバーコードと見なし分類データベースを作ります DNAバーコードの父と呼ばれるカナダ・ゲルフ大学の P. Hebert 博士によって提唱された概念です。生物種が異なれば、その生物の持っている遺伝情報、すなわち遺伝子DNAの4種類の塩基 (A, T, G, Cと略される) の並び方が異なります。したがって、特定の遺伝子に着目してその塩基配列(DNA バーコードとよばれる)を分析すれば、予め作成しておいたデータベースに照合することで、その生物種を特定することが可能になります。DNAの塩基配列を4色カラーで表示したものが、スーパーマーケットなどで商品の識別に使われるバーコードに似ているのでこのように呼ばれますが、生物の特定の遺伝子の塩基配列のことです。
DNA塩基配列を生物固有のバーコードと見なし分類データベースを作ります DNAバーコードの父と呼ばれるカナダ・ゲルフ大学の P. Hebert 博士によって提唱された概念です。生物種が異なれば、その生物の持っている遺伝情報、すなわち遺伝子DNAの4種類の塩基 (A, T, G, Cと略される) の並び方が異なります。したがって、特定の遺伝子に着目してその塩基配列(DNA バーコードとよばれる)を分析すれば、予め作成しておいたデータベースに照合することで、その生物種を特定することが可能になります。DNAの塩基配列を4色カラーで表示したものが、スーパーマーケットなどで商品の識別に使われるバーコードに似ているのでこのように呼ばれますが、生物の特定の遺伝子の塩基配列のことです。
今日、昆虫など様々な生物群における分類学専門家の数は減少し、後継者もなかなか育ちにくいという問題が世界的に存在します。DNAバーコーディングは、このような問題点を克服する一つの試みとも言えます。形態情報に基づく種同定は長年の経験に基づく熟練作業を要しますが、DNA塩基配列の決定とデータベースへの照合は、比較的簡便で統一的な作業で実現できるからです。動物では cox1 とよばれるミトコンドリアDNAの遺伝子、植物では matK、rbcL とよばれる葉緑体DNAの遺伝子を利用して、DNA バーコードのデータベースを作製しようというのが、DNA バーコード計画です。このプロジェクトに取り組む国際コンソーシアム(iBOL![]() )が2010年10月に作られ、全生物のDNAバーコードデータベースを作製する作業が進められています。2015年までの最初の5年間の目標は50万種でしたが、この目標は2015年8月に達成されたことが発表されました(2017年11月現在、58万種から530万バーコードを取得)。この技術は分類学だけでなく、環境保全、疫学、そして経済に様々な利益をもたらすことが期待されています。
)が2010年10月に作られ、全生物のDNAバーコードデータベースを作製する作業が進められています。2015年までの最初の5年間の目標は50万種でしたが、この目標は2015年8月に達成されたことが発表されました(2017年11月現在、58万種から530万バーコードを取得)。この技術は分類学だけでなく、環境保全、疫学、そして経済に様々な利益をもたらすことが期待されています。
日本以外の先進国の多く(カナダ、米国、EU7カ国、中国)は、iBOLにおいてCentral Nodesと呼ばれる主要な基幹国であり、その多くは国家プロジェクトとしてDNAバーコードデータの取得や解析、公開に主要な役割を果たしています。オーストラリア、ブラジル、インド、ロシア、南アフリカ共和国、韓国などの10カ国はRegional Nodesと呼ばれ、それらの国や周辺地域のDNAバーコーディングを積極的に推進しています。これ以外に、ケニアやパナマなど自分の国の生物多様性に絞って解析を担当するNational Nodesと呼ばれる国々も参加しています。2017年11月現在、残念ながら日本国のiBOLへの参加は実現していません。日本の研究者は、比較的小さなチームで個別に研究資金を獲得し、自らの得意とする分類群についてのDNAバーコーディングを行なっていますが(日本バーコードオブライフ・イニシアティブ![]() )、iBOL参加国の作業スピードには全く追いつかないのが現状です。我が国の生物多様性情報の取得が遅れるのは、基礎学問への影響にとどまらず、環境保全や生物資源・食品管理など国民生活への影響にも広く及ぶと考えられます。
)、iBOL参加国の作業スピードには全く追いつかないのが現状です。我が国の生物多様性情報の取得が遅れるのは、基礎学問への影響にとどまらず、環境保全や生物資源・食品管理など国民生活への影響にも広く及ぶと考えられます。
現在は、DNA塩基配列の決定に、実験室内でのDNA抽出、ポリメラーゼ連鎖反応(PCR)、ダイターミネーション反応、シーケンサーランなどのステップを要します。しかし、野外でこれらの実験を簡便にできる装置の開発も行われており、すでにプロトタイプの機器も発売されています。あと何年かすれば、アマゾンの奥地やアフリカの砂漠の中で、採集された生物試料のDNAバーコーディングが普通にできる時代が到来するかもしれません。
DNAバーコードの取得方法
試料採集
- 野外でサンプルを収集します。
- 分類の専門家が採集したサンプルの種を同定します。
DNA抽出
- 数ミリグラムのサンプルを取り、細片にします。
- タンパク質分解酵素と一緒に小さなチューブに入れて、しばらく加温してタンパク質を消化します。
- DNA抽出キットを用いて、DNAを抽出します。

メスを用いてサンプル細切しているところ。

サンプルを消化中
DNA増幅 (PCR)
- 抽出したDNAをもとに、目的とするDNA断片をPCR法により増幅します。
- DNAが増えていることをゲル電気泳動により、確かめます。
DNAの電気泳動
塩基配列分析
- DNAに蛍光で目印をつける反応を行い、DNA塩基配列分析装置で分析します。当センターには、Thermo Fisher Scientific社の3500 Genetic Analyzer(8本キャピラリーシーケンサー)が設置されています。

DNA塩基配列分析装置
DNAデータベースに登録
- DNAバーコード情報(学名、産地、名市大の標本庫などにおける標本登録番号、標本画像、DNA塩基配列、シーケンスを読み取ったときの継時的な蛍光パターンの図など)は、BOLDシステム
 に登録します。DNAの塩基配列から種を推定しようとする場合も、このデータベースで既存データに照合します。
に登録します。DNAの塩基配列から種を推定しようとする場合も、このデータベースで既存データに照合します。
![]()
ACGTの4種類の塩基を色分けしたもの
DNAバーコードの例
よく似た葉をもつ植物でも、DNAを調べることにより、種を同定できるだけでなく、お互いにどれとどれが近縁種かということもわかります。実際に、木曽五木のうち葉の形のよく似たの4種の樹木のDNAバーコード (matK 遺伝子) を調べてみました。

アスナロはヒノキよりもネズコにちかいのでしょうか。DNAを調べた結論は、アスナロはなかなかヒノキになれないかもしれません。
DNA分析により、植物の分類も見直しが行われている
形態学的は双子葉植物から単子葉植物が進化したと考えられていましたが、多くの植物の遺伝子DNAが解析されて、単子葉植物が現れる以前にもモクレンのような原始的な双子葉植物があらわれたと考えられるようになってきました。
進行中のDNAバーコード計画
日本産貝類のDNAバーコーディングと系統解析
 海洋国家日本の近海には、6000種とも言われる多様な貝類が生息しています。また、その一部は食用にもなるなど、市民に馴染み深い種も含まれます。また陸域や淡水域にも1000種以上とも言われる多様な貝類が生息しています。長期目標の一つは日本産貝類全種のDNAバーコードデータベースの構築ですが、まずは東海地方の陸産、淡水産の貝を中心にDNAバーコードデータの収集・分析及び系統解析を開始しました。センター研究員の川瀬基弘博士(愛知みずほ大学講師)が収集した貝類標本を主に用いており、研究後の標本は標本庫に預け入れられています。今後、海産の種にも研究対象を広げていく予定です。
海洋国家日本の近海には、6000種とも言われる多様な貝類が生息しています。また、その一部は食用にもなるなど、市民に馴染み深い種も含まれます。また陸域や淡水域にも1000種以上とも言われる多様な貝類が生息しています。長期目標の一つは日本産貝類全種のDNAバーコードデータベースの構築ですが、まずは東海地方の陸産、淡水産の貝を中心にDNAバーコードデータの収集・分析及び系統解析を開始しました。センター研究員の川瀬基弘博士(愛知みずほ大学講師)が収集した貝類標本を主に用いており、研究後の標本は標本庫に預け入れられています。今後、海産の種にも研究対象を広げていく予定です。
関連して発表した論文をいくつか以下に示します。
【成果発表】
●熊澤慶伯,松原美恵子,横山悠理,寺本匡寛,村瀬幸雄,那須健一郎,孫垚,森山昭彦,川瀬基弘(2019)名古屋市産淡水貝類のDNAバーコーディング. なごやの生物多様性 6: 1-14. ![]() 論文PDF
論文PDF
●川瀬基弘,西尾和久,松原美恵子,市原俊,森山昭彦,熊澤慶伯(2018)遺伝子解析に基づく中部・西日本産ナミギセルStereophaedusa japonica個体群の種内多様性と名古屋市の個体群の系統的位置づけ. なごやの生物多様性 5: 11-22. ![]() 論文PDF
論文PDF
●川瀬基弘,松原美恵子,森山昭彦(2016)愛知県西三河地域から採集されたヒラマキガイ属3種:形態と遺伝子情報による解析. 陸の水 74: 43-48.
●川瀬基弘,西尾和久,松原美恵子,森山昭彦(2016)新城市のミカワギセルの特徴とCOI遺伝子からみた分子系統関係. 鳳来寺山自然科学博物館館報 (45): 27-30.
●川瀬基弘,西尾和久,森山昭彦,市原 俊,桜井栄一(2015)ミヤマヒダリマキマイマイ(腹足綱: ナンバンマイマイ科)種内の形態変異と分子系統. Molluscan Diversity 4(1-2): 5-13.
コメツキムシのDNAバーコーディング
 写真提供:大場裕一博士コメツキムシは、日本に広く存在する甲虫で、ホタルのように光る種類もあります。世界には約1万種が、日本でも約600種(その多くは日本の固有種)が知られています。一部のコメツキムシは農業害虫にもなっており、幼虫の種同定技術への期待は高いのですが、形態に基づく同定法には一定の限界がありました。多数の昆虫愛好家のご協力を得て収集した成体標本(275種、762個体)を用い、コメツキムシの分類学者(大平仁夫氏)による形態に基づく種同定の結果とDNAバーコードによる種境界の認識結果を比較しました。その結果、275種のうち177種については、形態と分子に基づく種境界が一致しました。一方、形態に基づき同定された47種は、実際には184種を包含する可能性があることを分子データは示唆しました。本研究は、日本の野外で普通に見られるコメツキムシを専門家の鑑定なしで同定できるDNAバーコードデータベースを構築したことに加え、コメツキムシの分類学上の検討課題をいくつか提示した意義があると考えます。
写真提供:大場裕一博士コメツキムシは、日本に広く存在する甲虫で、ホタルのように光る種類もあります。世界には約1万種が、日本でも約600種(その多くは日本の固有種)が知られています。一部のコメツキムシは農業害虫にもなっており、幼虫の種同定技術への期待は高いのですが、形態に基づく同定法には一定の限界がありました。多数の昆虫愛好家のご協力を得て収集した成体標本(275種、762個体)を用い、コメツキムシの分類学者(大平仁夫氏)による形態に基づく種同定の結果とDNAバーコードによる種境界の認識結果を比較しました。その結果、275種のうち177種については、形態と分子に基づく種境界が一致しました。一方、形態に基づき同定された47種は、実際には184種を包含する可能性があることを分子データは示唆しました。本研究は、日本の野外で普通に見られるコメツキムシを専門家の鑑定なしで同定できるDNAバーコードデータベースを構築したことに加え、コメツキムシの分類学上の検討課題をいくつか提示した意義があると考えます。
本研究は、名古屋大学生命農学研究科(現中部大学)の大場裕一先生![]() と当センターの共同研究で、この成果は2015年にPLoS ONE誌に論文発表されました。また、日本農業新聞でも記事として紹介されました(2015年3月19日付)。
と当センターの共同研究で、この成果は2015年にPLoS ONE誌に論文発表されました。また、日本農業新聞でも記事として紹介されました(2015年3月19日付)。
【成果発表】Oba Y, Ôhira H, Murase Y, Moriyama A, Kumazawa Y (2015) DNA barcoding of Japanese click beetles (Coleoptera, Elateridae). ![]() PLoS ONE 10(1): e0116612. doi:10.1371/journal.pone.0116612
PLoS ONE 10(1): e0116612. doi:10.1371/journal.pone.0116612
![]() 日本バーコードオブライフ・イニシアチブ(日本産コメツキムシ)
日本バーコードオブライフ・イニシアチブ(日本産コメツキムシ)![]() コメツキムシDNAバーコードデータベースへのリンク
コメツキムシDNAバーコードデータベースへのリンク
ゾウムシのDNAバーコーディング
コクゾウムシの類は、クチ(口吻)が長いのでその形から「ゾウムシ」と呼ばれます。種々の植物に対応していろいろなゾウムシがおり、環境の豊かさの指標になります。名古屋市内だけでも100種を越えるゾウムシが見つかっています。体長数mmと小型のものが多く、よく似ていているので外見だけで種類を決めることはなかなか困難です。名古屋市を中心とした東海地方のゾウムシ類(85種、164個体)を解析した結果について、下記の原著論文を発表しました。土壌性のゾウムシ類などで、愛知県の山ごとに別種に分化している可能性が示されました。また、この研究成果を含めて、「なごやの甲虫とゾウムシの多様性」のテーマで、第5回なごや生物多様性センターまつりに出展しました(2018年10月27日)。
【成果発表】井上晶次、熊澤慶伯 (2017) 名古屋市を中心とした愛知県及び近隣県産ゾウムシ類のDNAバーコーディング.なごやの生物多様性 4:23-29. ![]() 論文PDF
論文PDF
![]() 出展ポスターのPDF
出展ポスターのPDF
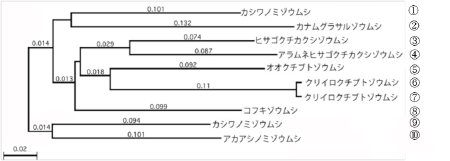
以下は、9種類のゾウムシを分析した例です。 写真提供:井上晶次氏
写真提供:井上晶次氏
なごや生物多様性保全活動協議会との連携
なごや生物多様性保全活動協議会(通称なごビオ)が毎年行っている「なごや生きもの一斉調査」で確認された生物についてさらに詳しく分析するため、DNA等を用いた調査研究を行っています。2017年度に実施した名古屋市内の淡水貝類の一斉調査で得られた標本については、約300点の標本を名市大の標本庫へ寄贈して頂き、DNAバーコーディングの研究に用いました。その他、昆虫類や両生類などに関しても協議会のメンバーとコラボした研究を実施しています。
【成果発表】
●熊澤慶伯,松原美恵子,横山悠理,寺本匡寛,村瀬幸雄,那須健一郎,孫垚,森山昭彦,川瀬基弘(2019)名古屋市産淡水貝類のDNAバーコーディング. なごやの生物多様性 6: 1-14. ![]() 論文PDF
論文PDF
東山総合公園の動植物のDNAバーコーディング
 東山植物園のヤブツバキ 東山動植物園では、動植物そのもの、あるいはその生態への理解を深めていただくために様々な動植物を展示しています。展示されている動植物について、学名や生息地だけでなく、遺伝子情報も把握しておくことは、動植物園としては重要なことです。その理由の一つを挙げますと、動物園にいるゾウ、ゴリラ、ワニなどの絶滅危惧種を絶やさないためには、国内外の動物園等にいる同種(同亜種)個体と遺伝情報に基づいて適切に交配させる必要があるからです。私たちは動植物園と協力して試料を収集するとともにDNA塩基配列を調べています。近縁種が数多く保存されている動植物種では、それらの遺伝的類縁関係について明らかになってくることも期待されています。
東山植物園のヤブツバキ 東山動植物園では、動植物そのもの、あるいはその生態への理解を深めていただくために様々な動植物を展示しています。展示されている動植物について、学名や生息地だけでなく、遺伝子情報も把握しておくことは、動植物園としては重要なことです。その理由の一つを挙げますと、動物園にいるゾウ、ゴリラ、ワニなどの絶滅危惧種を絶やさないためには、国内外の動物園等にいる同種(同亜種)個体と遺伝情報に基づいて適切に交配させる必要があるからです。私たちは動植物園と協力して試料を収集するとともにDNA塩基配列を調べています。近縁種が数多く保存されている動植物種では、それらの遺伝的類縁関係について明らかになってくることも期待されています。
2010年から試料の収集と分析を始め、2015年までに102種118個体の脊椎動物でcox1遺伝子の配列決定と解析を行いました。また、221種の植物でrbcL遺伝子を、159種の植物でmatK遺伝子の配列決定と分析を行いました。現在までに以下の3点の報告書を作成しています。それらの報告書からの抜粋をポスターにして、第4回なごや生物多様性まつりで東山動植物園と共同展示を行いました。
|
東山公園DNAバーコードプロジェクト報告書(2013年3月)
|
|
|
東山植物園「ハスとスイレンのDNAバーコーディング」報告書(2015年4月)
|
|
|
東山動物園「DNAバーコードプロジェクト」報告書(2016年7月)
|
名古屋圏の植物の葉・DNA収集
名古屋市環境局なごや生物多様性センターでは、名古屋圏の植物に関する生息調査を行うかたわら、植物標本の収集と収蔵庫への登録を進めています。中には東海丘陵要素植物群のような東海地方固有の植物や名古屋市において希少種となった植物も含まれます。ただ現状ではDNA解析に対応した収蔵環境が実現できていないため、名市大の標本庫が葉標本の提供を受け、DNA分析に対応した遺伝資源の保存を行なっています。まだこの事業は始まったばかりでDNAデータも蓄積していませんが、将来的な植物種の保全や持続的利用に向けた貴重なリソースとなることが期待されます。


